
Result
02

地球上の生物が持つタンパク質は、L型/D型の2種類が存在するアミノ酸のうち、L型によって構成されており、D型は一部の細菌の細胞壁に存在するのみであると考えられてきました。しかし近年、哺乳類にもD型アミノ酸が存在することが明らかになるとともに、D型アスパラギン酸が、タンパク質の異常な折り畳みが原因で生じるヒトの疾病(アルツハイマー病や白内障)の病変組織や、皮膚の老化した組織に存在することが報告されました。一方、これまでD型アミノ酸を特異的に認識するペプチダーゼの報告はほとんどなされておらず、とりわけD型アスパラギン酸特異的エンドペプチダーゼ(PAE)は、微生物由来の酵素としては世界で唯一の例です。
私たちはこれまでに、新規D-アスパラギン酸基質を合成し、D-アスパラギン酸特異的エンドペプチダーゼ生産菌(Paenibacillus sp. B38株)を分離するとともに、産生する酵素をpaenidase (PAE)と命名し、その性質を解析しました(S. Takahashi et al., J Biochem, 2006)。続いて、PAE遺伝子を同定し、大量発現系を構築するとともに、PAEの活性部位をアミノ酸変異法により推定しました。また、PAEにより分解したペプチドの分析方法や(H. Maeda et al., Anal Chem, 2015)、慢性閉塞性肺疾患の病変組織にD-アスパラギン酸が含まれることを報告しており(M. Ogasawara et al., Exp Lung Res, 2016)、PAEがD型アスパラギン酸の検出ツールとして利用価値が高いことを明らかにしてきました。
このように細胞生物学・生化学的な知見が蓄積する一方、PAEが活性を発揮する構造基盤についてはこれまで全く明らかになっていませんでした。とりわけ、PAE がD型アスパラギン酸を認識する機構については、他に比較する対象がなく、まったく不明でした。類似酵素等との配列相同性の情報やホモロジーモデリングによって、基質認識部位の探索も行いましたが、当該部位の同定には至りませんでした。そのため、PAEの結晶構造解析を実施し、活性部位および基質認識部位の精密な立体構造を決定することで、酵素活性を発揮するメカニズムの解明に結びつけたいと考え、過去に別の共同研究によって結晶構造解析を目指したこともありましたが、残念ながら構造決定には至りませんでした。
その後、PAEの構造解析に関する研究は中断していましたが、日本農芸化学会2016年度大会(2016年3月27日?3月31日。於:札幌)でJAXAの方とお話ししたことが縁で、JAXA高品質タンパク質結晶生成実験に参加することになりました。その際、私たちのグループ内にはX戦結晶構造解析の専門家はいなかったため、共同研究の役割分担として、私たちのグループではタンパク質試料と基質・阻害剤の提供をし、JAXA側で結晶化条件の探索および構造解析を実施するということになりました。
共同研究開始後、幸いにも数ヶ月で新たな結晶化条件が見つかり、2017年2月には最初の宇宙実験(LTPCG#1)を実施しました。その実験で、約2.25Aの分解能を示す高品質結晶を取得することに成功し、世界で初めて、「D型アスパラギン酸特異的エンドペプチダーゼ(PAE)」の構造を明らかにしました(図1)。地上実験で得られた結晶では、最高分解能が約3.5Aであったことから、宇宙実験による結晶の品質向上がタンパク質の精密構造解析に寄与したことになります。また、宇宙実験で得られたPAEの結晶に、D型アスパラギン酸特異的エンドペプチダーゼ阻害剤であるi-DAEP(Bz-Arg-His-[D-Asp]-CH2Cl)をソーキングし、酵素阻害剤複合体結晶を取得しました。得られた複合体結晶を解析したところ(図2A)、i-DAEPのD-Asp残基の側鎖と、PAEのArg121の側鎖が結合していることが明らかになりました(図2B)。
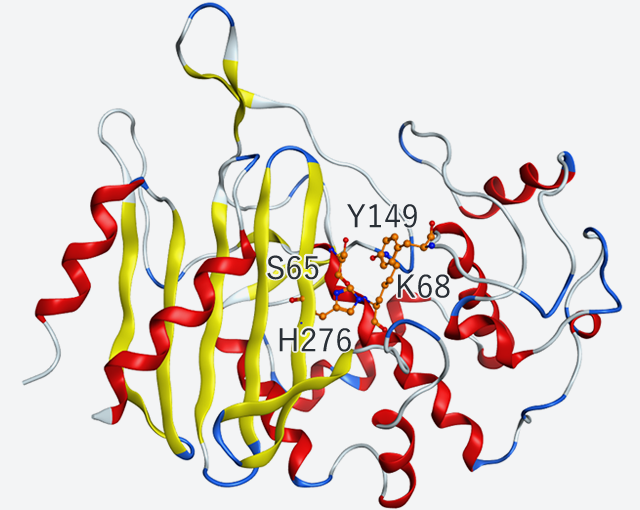
図1
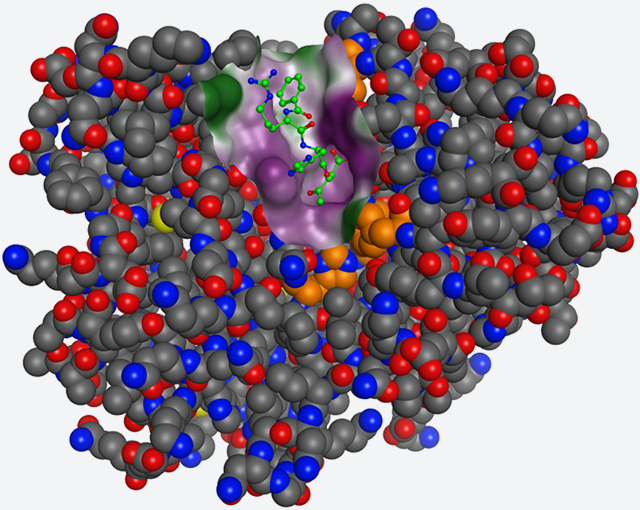
図2(A)
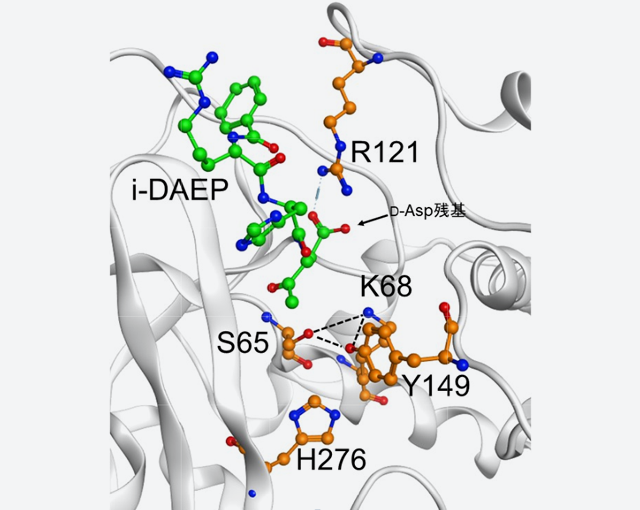
図2(B)
図1. D型アスパラギン酸特異的エンドペプチダーゼ(PAE)の全体構造及び活性残基
図2. D型アスパラギン酸特異的エンドペプチダーゼ(PAE)と阻害剤i-DAEPとの複合体の構造
(A) 複合体の全体構造。i-DAEPは緑色で示されている。Bz基とCl原子は非表示。
(B) PAEの121番目のArg残基(R121)の側鎖と、i-DAEPのD-Asp残基の側鎖が結合している。
これまでの研究で、PAEは細菌類がもつ細胞壁合成酵素群と似ている点があることがわかっていたので、今回解析したPAEの構造と細胞壁合成酵素群の構造を重ね合わせ、比較してみました(図3)。その結果、酵素の活性部位であるSer、Lys、Tyr、Hisの位置は良く一致する一方、PAEの基質認識部位であるArg121を含むループ部分の構造はそれぞれの酵素で大きく異なることが明らかになりました。以上のことから、これら酵素の基質特異性の違いは、ループ部分に含まれるアミノ酸残基の種類と立体配置に起因すると考えるに至りました。
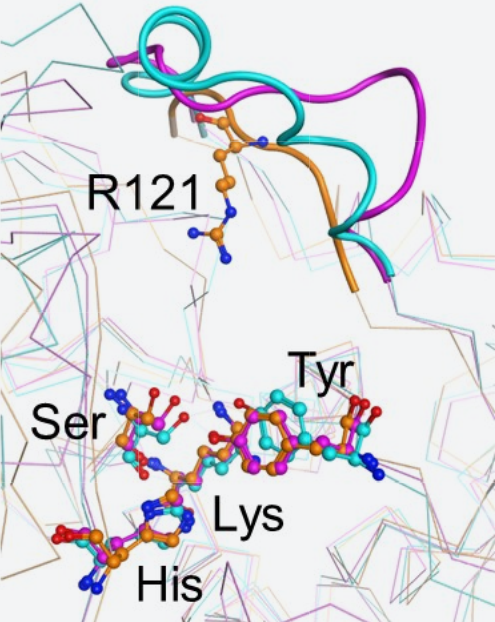
図3
図3. D型アスパラギン酸特異的エンドペプチダーゼ(PAE)と細胞壁合成酵素群の構造の重ね合わせ
橙色:PAE
水色:D-Ala-D-Ala carboxypeptidase B(Streptomyces strain R61由来)
桃色:Alkaline D-peptidase(Bacillus cereus由来)
Ser、Lys、Tyr、Hisは活性部位を示す。ループ部分は太線で示した。
つぎに、基質認識部位Arg121を各種アミノ酸(Ala, Ile, Glu, His, Lys)に変異させ、それら(R121A, R121I, R121E, R121H, R121K)の酵素活性を解析しました。その結果、Argと同じ塩基性アミノ酸であるLysに置換した場合(R121K)では、約44%の活性を保持しました(図4)。一方、Ala, Ile, Glu, Hisに置換した場合では、活性が著しく低下しました(図4)。
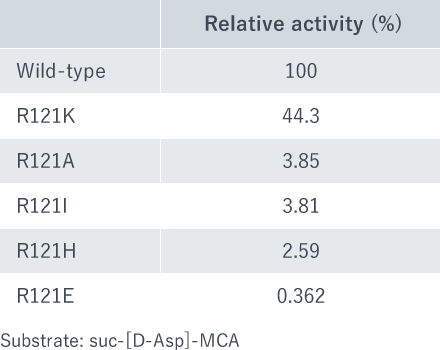
図4
図4. 各種変異酵素の相対活性
基質として、suc-[D-Asp]-MCAを用いた。
したがって、基質結合には、側鎖の正電荷が重要であることが明らかになりました。さらに、アミロイドβタンパク質 (Aβ)のN末端から10残基目までのペプチドを用いて、酵素活性を解析しました。このペプチドでは、7番目のAsp残基がD型に置換されています。図5に示すように、Wild-typeでは、7番目のD-Asp残基のC末端側が切断され、2つのペプチドに完全分解されました。一方、面白いことに、変異酵素R121K、R121Aでは、ペプチドを完全には分解できませんでした。したがって、ペプチドが基質の場合、ArgとLysの側鎖の立体配置の違いが、酵素の反応速度に大きく影響すると考えられます。
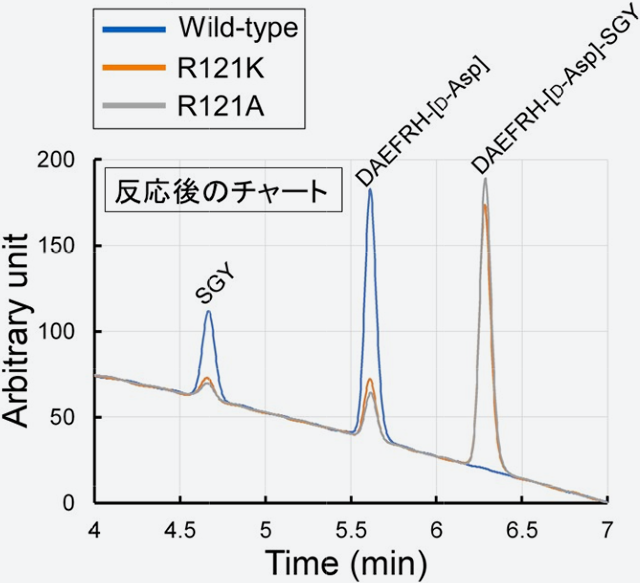
図5
図5. 各種変異酵素によるD型Asp含有Aβデカペプチドの分解
酵素反応後、高速液体クロマトグラフィー(HPLC)により分析した。Wild-type(青色の線)では、2つのペプチド(DAEFRH-[D-Asp]、及びSGY)に完全分解したが、変異酵素(橙色及び灰色の線)では、ほとんど分解されていない。
このように、なかなか進まなかったPAEの基質認識機構の理解ですが、立体構造解析を契機として著しい進展を見せました。今後は、得られた知見を活用し、新規抗菌剤の検索や阻害機構の解析を推進するとともに、アルツハイマー病や白内障などのD型アスパラギン酸の検出を基盤とした疾病診断法への応用展開につなげていきたいと考えています。

韮澤悟 主任研究員